Uno de los descubrimientos más intrigantes de la era genómica es que solo una pequeña fracción del genoma está dedicada a la codificación de proteínas. La fracción restante de él contiene, entre otros elementos, una serie de transcritos no codificantes que regulan la transcripción de genes codificadores de proteínas.
En ese sentido, un estudio de Investigadores de la Agencia de Investigación Aplicada de Australia, (CSIRO, por su sigla en inglés), en colaboración con el Centro Interdisciplinario para la Investigación Acuícola (INCAR), demuestra el importante rol de los ARNs no codificantes en la regulación de la expresión de genes asociados a procesos infectivos en salmón del Atlantico.
La investigación liderada por la Dra. Francisca Samsing (CSIRO), junto a Dr. James W. Wynne (CSIRO), y la Dra. Pâmela A. Alexandre (CSIRO), en colaboración con Dra. Valentina Valenzuela-Muñoz (INCAR), Dr. Diego Valenzuela-Miranda (INCAR), Dr. Cristian Gallardo-Escárate (INCAR), describe la respuesta molecular de tejido branquial de salmón del Atlántico (Salmo salar) infectado con el ortomixovirus de la sardina (POMV), mediante redes de interacción mRNA-miRNAs.
Los científicos examinaron dos grupos de peces infectados por cohabitación con el virus POMV: grupo de exposición temprana 8 a 12 días después de la infección (dpi), para identificar potenciales biomarcadores de infección temprana e inmunidad innata. Lo que fue comparado con peces de exposición tardía, 19 dpi, para dilucidar potenciales marcadores de resistencia a POMV. Para ello, realizaron secuenciación de transcriptoma. De manera de identificar ARNs mensajeros expresados diferencialmente e identificar nuevos ARNs no codificantes largos (lncRNAs). Secuenciacion de smallRNAs encontrar microARNs diferencialmente expresados. Adicionalmente, mediante analisis de co-expresión y redes se pudo determinar genes y lncRNAs potencialmente modulados por miRNAs diferencialmente expresados durante la infección viral.
Los investigadores observaron que el grupo de microRNAs, miR-462/miR-731 específico de teleósteos, es altamente inducido en peces infectados con ortomixovirus. Además, se sugiere el posible uso de estos miRNAs como potencial biomarcador de infección viral temprana. Por otra parte, se observó disminución de la expresión del gen selenoproteína (selja), responsable del transporte de selenio, en peces de infección tardía (19 días post-infección). Los autores discuten que el cambio de expresión de este gen puede estar asociada con la eliminación viral y el regreso a la homeostasis después de la infección. Finalmente, se sugiere que la proteína selja tendría un rol relevante en peces durante una infección viral.
Revisa el paper “Competing endogenous RNA-networks reveal key regulatory microRNAs involved in the response of Atlantic salmon to a novel orthomyxovirus”, publicado en el Journal Developmental & Comparative Immunology.
https://doi.org/10.1016/j.dci.2022.104396
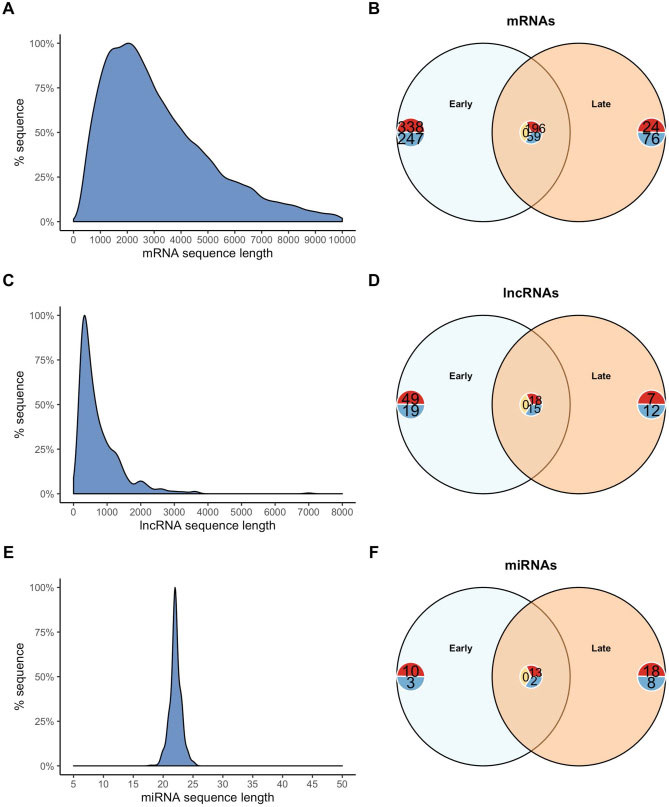
Figura 1: Características y perfiles de expresión génica del huésped de transcripciones codificantes (ARNm) y no codificantes (ARNlnc y miARN) en salmón del Atlántico subclínico (Salmo salar) desafiado con el ortomixovirus de la sardina (POMV). Distribución de longitud para (A) todos los ARNm ensamblados; (C) lncRNA predicho; y (E) miARN anotados. Diagramas de Venn para ARNm expresado diferencialmente (B); (D) lncRNA; y (F) miARN.
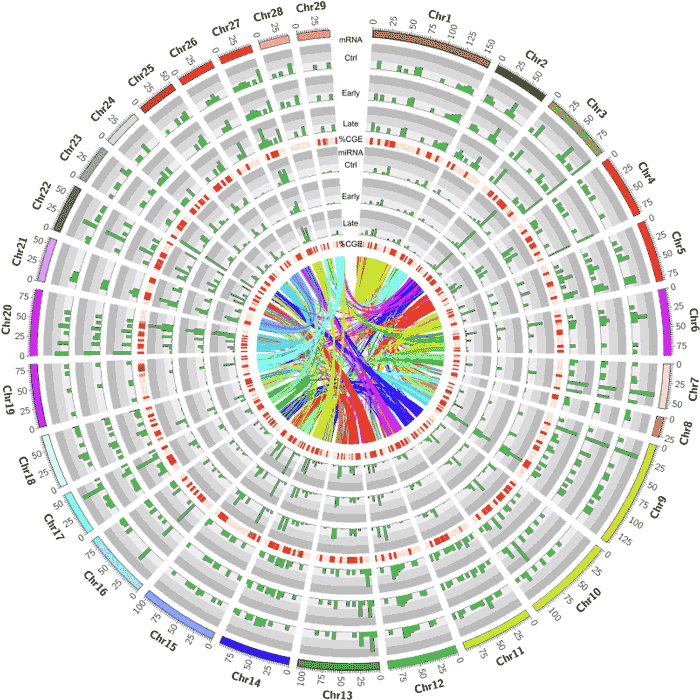
Circulogenoma: Transcripción del genoma completo en salmón del Atlántico infectado con POMV. El mapa de calor en rojo muestra la variación del índice de expresión del genoma cromosómico (CGE) entre el grupo de peces con respuesta temprana y tardía a la infección por POMV. Las cintas, en el diagrama de Circos, representan las regiones homólogas en el genoma del salmón.